type
status
date
slug
summary
tags
category
icon
password
Property
Mar 6, 2024 11:23 AM
Created time
Jan 31, 2024 05:37 PM
说明:本文为《数据挖掘》课程期末实验,实验要求难度非常大,实验引导极不充分。实验期间本人又感染新冠未愈,所以文章质量很差,属于是应付作业,请酌情斟取。
一、实验目的
- 了解深度矩阵分解的目的,内容以及流程
- 对给定数据集进行深度矩阵分解,并进行可视化
- 掌握深度矩阵分解,编程实现合成致死数据集的关系预测
二、实验过程及结果分析
2.1 数据预处理
本次实验参考了开源项目deep_matrix_factorization的实现,该开源项目和现有论文大都是将深度矩阵分解用作推荐系统,所使用数据集矩阵以用户为行,以电影编号/商品编号为列,以该用户对电影/商品的评分作为矩阵值。
本次作业中老师提供的数据集又一次不做说明,严重影响了实验工作的开展。参考相关论文信息,我大致了解到:
- gene_list.txt文件保存了数据集中涉及到所有基因名称;
- Gene_GO_similarity.txt文件记录的是基因之间GO数据语义相似度的邻接矩阵;
- Gene_PPI_similarity.txt文件保存的是基因编码蛋白质间的拓扑相似度邻接矩阵;两文件均未说明各自数据的数据来源、计算方法和保存规则,我只能简单地认为邻接矩阵的行列均按照gene_list.txt文件内基因名排序进行。
- Human_Sls.txt文件保存了目前已知的合成致死基因对,每个基因对后紧随的数值为该基因对为合成致死基因对的置信度。
我不知道相似度文件内保存值的计算方法与量纲,譬如Gene_GO_similarity和Gene_PPI_similarity中每一个基因对自己本身的相似度均为0,这是很奇怪的。
这里按照参考开源项目的数据集设置,我将Human_Sls.txt文件中的基因名称按照gene_list.txt中的顺序均进行数字编号映射,并且将置信度数值扩大10倍转为整数。
预处理后的数据如下图所示,原本U/I/Q分别代表用户,商品编号和评分,这里代表基因1编号、基因2编号、该基因对是合成致死基因对的置信度。待分解的初始矩阵大小为U*I,这里直接设为基因总数6375 * 6375。
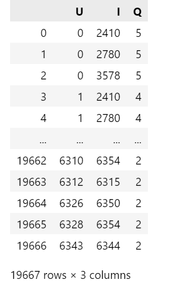
随后将数据打乱,分成训练数据、验证数据和测试数据
2.2 深度矩阵分解
2.2.1 算法原理
矩阵分解(matrix factorization, 简称MF)就是把原来的大矩阵,近似的分解成小矩阵的乘积,在实际推荐计算时不再使用大矩阵,而是使用分解得到的两个小矩阵。具体来说就是:假设用户物品的评分矩阵A是m乘n维,即一共有m个用户,n个物品.通过一套算法转化为两个矩阵U和V,矩阵U的维度是m乘k,矩阵V的维度是n乘k。这两个矩阵的要求就是通过下面这个公式可以复原矩阵A: 而直观上说U矩阵就是m个用户对k个主题的关系,Q矩阵就是K个主题对M个物品的关系,至于说K个主题具体是矩阵分解算法里的一个参数,一般取10到100之间。
深度矩阵分解通过在传统矩阵分解的基础上,加入神经网络相关的隐藏层,企图让分解结果包含有更多隐式信息和用户与商品数据间非线性的关系,下面是两种主要的算法示意图,本实验中的算法实现更加接近下方右图。
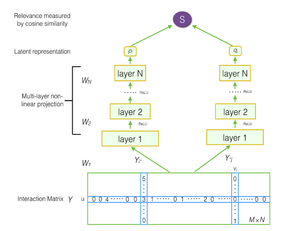
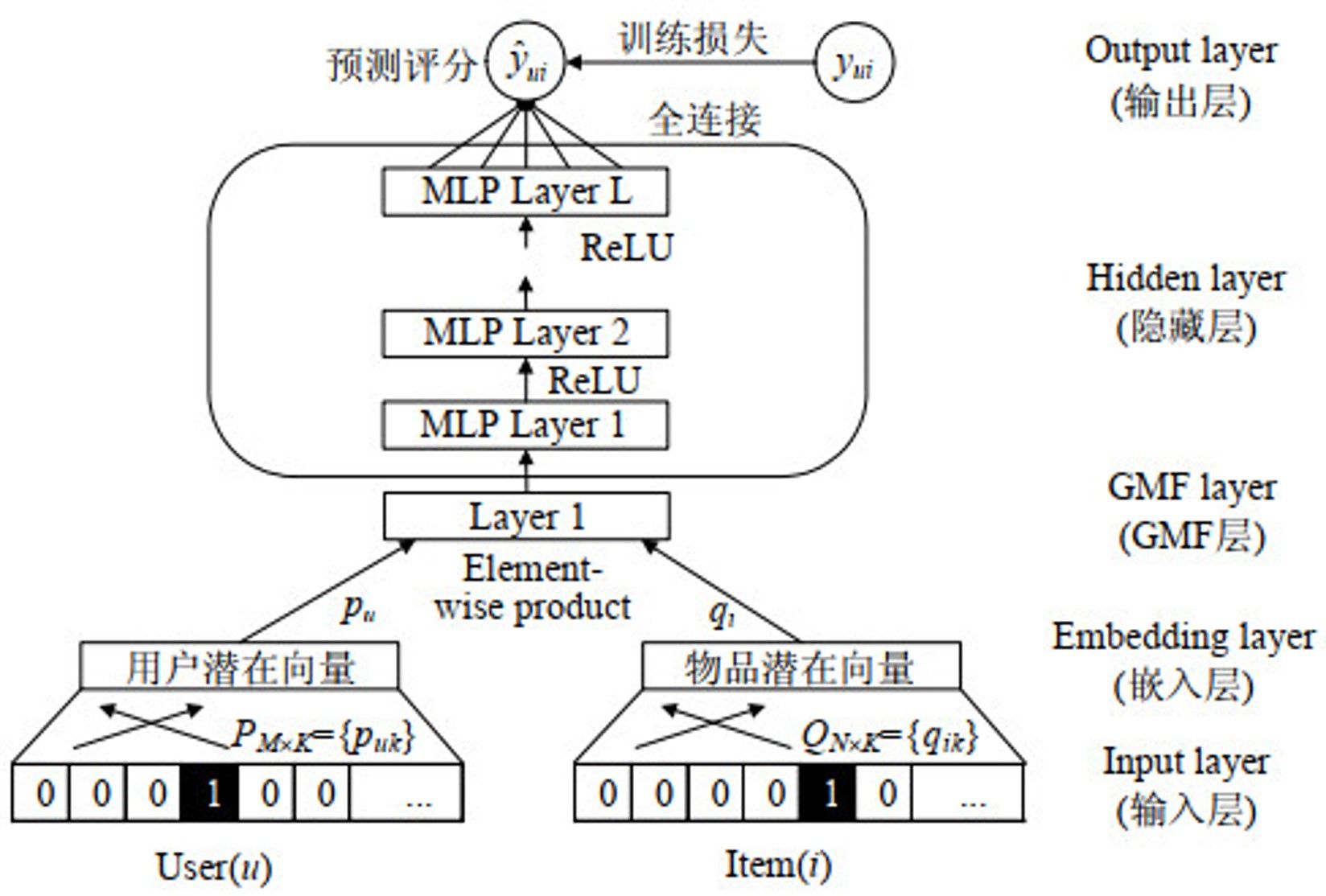
2.2.2 算法实现
下面参照开源项目,实现了一个深度矩阵分解类,使用到tensorflow和numpy。
- 初始化
初始化构造函数展示了整个模型的大体配置,原始输入矩阵将会被分解为u,i两个部分,依次经过嵌入层和多个隐藏层的处理得到输出,这里使用最简单的均方差MSE作为损失函数用以评估
- 训练函数
fit函数将对输入的训练数据分批导入到TensorFlow实例中用以训练
- 预测函数
predict函数将调用训练得到的output模型,将x作为输入,x一般是[用户,商品]对,在本实验中为基因对,输出结果为用户对该商品的评分预测,本实验中为该基因对为合成致死基因对的置信度。
evaluate函数将会比较现有模型对输入x的预测结果和实际结果y的差异,计算方差和。
2.2.3 模型实例化
创建一个深度矩阵分解模型实例,导入前面预处理好的数据,设置50次迭代,通过绘图比较损失函数的变化,结果如下:
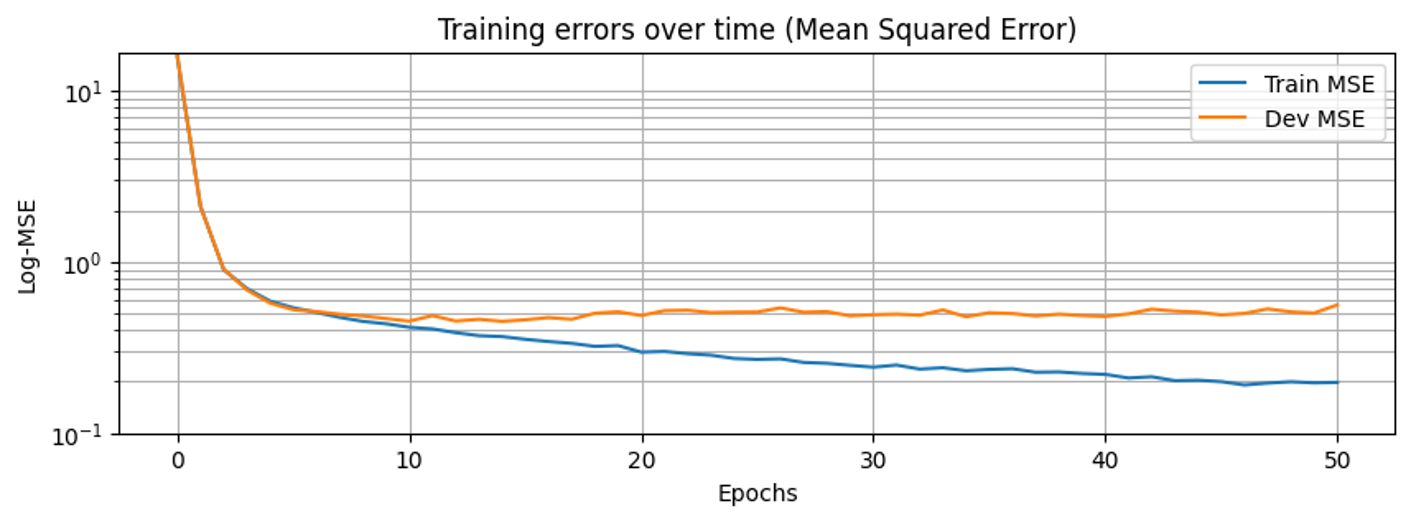
可以看到训练数据的损失函数值逐渐收敛,但是验证数据的损失却又开始回升,这是数据过拟合的表现,适当调整参数可能会有所改善。
测试数据的结果
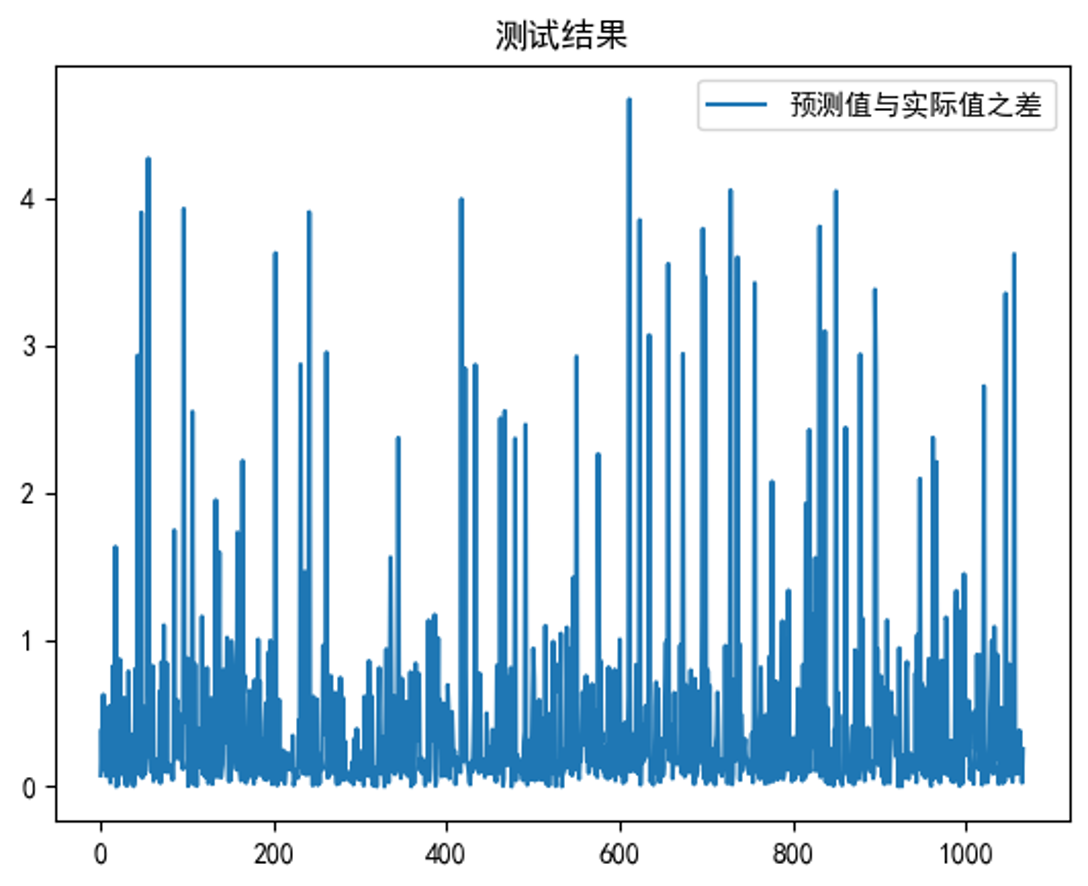
可以看到,测试结果偏差大都在0-1之间,整体结果还可以。
2.2.4 模型用于合成致死数据集的关系预测
对所有基因对进行预测,并按照评分值(对应原先矩阵中的置信度)大小进行降序排名,保留前一百个结果。
首先构造所有的基因对,这里为了减少计算时间,只取基因对矩阵U-I中的右上对角线一侧数据
接着使用训练好的模型进行预测,因为预测的基因对数量较大,我这里运行了将近5分半钟,如果左下对角线的基因对数据也进行预测的话在本人机器上的运行时间大致在9到10分钟。
随后对评分结果进行排序
打印出评分最高的前100个基因对信息如下
打印信息较长,完整内容可在源代码中进行查阅。

可以观察到,CYLD,C7ORF50, CYP11B1这三个基因在前一百高评分基因对的左侧出现得非常频繁,而在原始数据集给出的合成致死基因对中这三者的数据量并不大,因此从本次实验模型的预测结果来看,可以重点关注以这三个基因为代表的相关基因对并开展研究,可能对进一步发现和了解新的合成致死基因对有帮助。
2.2.5 模型的优化方向
对于矩阵分解本身来说,损失函数的定义对模型的性能有着较大影响,本实验中只是简单地使用均方差进行损失优化,仍具有较大的优化空间。
在论文《Deep Matrix Factorization Models for Recommender Systems》中使用的是交叉熵相关的数据作为损失函数,但是在论文相关项目的复现(https://github.com/RuidongZ/Deep_Matrix_Factorization_Models/issues/3)中,也有人提出这一选择在推荐系统中的不合理性。
在论文《SL2MF: Predicting Synthetic Lethality in Human Cancers via Logistic Matrix Factorization》中,文章使用的损失函数结合了蛋白质-蛋白质相互作用(PPI)数据和基因本体论(GO)的数据。我也尝试复现该损失函数的相关配置,但是调整代码多次后仍未成功运行,可能是对TensorFlow的理解仍不清晰导致的,考虑到个人身体原因,作业截止日期亦已临近,就暂且作罢,没有更加深入地研究下去了。
三、实验总结
通过本次实验我研读了矩阵分解研究方向的许多论文,并亲自上手实现深度矩阵分解,加深了使用矩阵分解的推荐/预测系统的原理理解,但是也暴露了在深度学习相关工具上运用不熟练的问题,需要在今后进一步加强。
- 作者:叶修齐
- 链接:https://notion.siuze.top/article/c0079e1f-b152-4a42-822e-a9666fbdbc92
- 声明:本文采用 CC BY-NC-SA 4.0 许可协议,转载请注明出处。
相关文章